1. 概述
本文档主要用于指导用户在使用曦云® 系列GPU时,如何安装部署和应用mcDF。
1.1. mcDF介绍
mcDF是一个基于曦云系列GPU,运算、处理DataFrame数据对象的Python计算库。 mcDF提供包括加载、连接、聚合、筛选以及其他数据操作接口,提供的API接口类似pandas对应功能的API接口,熟悉pandas库的数据工程师可以轻松地将基于pandas的Python应用迁移到mcDF上。
mcDF-c是mcDF的C++版本,包含一系列动态链接库,覆盖mcDF大部分的功能和操作接口。
Dask-mcDF允许在分布式GPU环境中进行高性能的数据处理。
1.2. 软件包
1.2.1. 软件包信息
mcDF提供了mxc500-mcdf-<VERSION>-linux-x86_64.tar.xz,可以使用命令 tar -xJf 进行解压,解压后可获得依赖的mcPy二进制软件包和mcDF二进制软件包。
mcPy二进制软件包的说明,参见《曦云® 系列通用计算GPU mcPy使用手册》。mcDF二进制软件包的信息说明,参见表 1.1。
软件包类型 |
文件名示例 |
说明 |
|---|---|---|
Python wheel包 |
mcdf-x.y.z.n+bu.v.w.m-cp38-cp38-linux_86_64.whl |
适用于Python 3.8的mcDF Python二进制扩展包 |
Python wheel包 |
mcdf-x.y.z.n+bu.v.w.m-cp310-cp310-linux_86_64.whl |
适用于Python 3.10的mcDF Python二进制扩展包 |
Python wheel包 |
mcpy-x.y.z.n+bu.v.w.m-cp38-cp38-linux_86_64.whl |
适用于Python 3.8的mcPy Python二进制扩展包 |
Python wheel包 |
mcpy-x.y.z.n+bu.v.w.m-cp310-cp310-linux_86_64.whl |
适用于Python 3.10的mcPy Python二进制扩展包 |
Python wheel包 |
numbax-x.y.z.n+bu.v.w.m-cp38-cp38-linux_86_64.whl |
适用于Python 3.8的numbax Python二进制扩展包 |
Python wheel包 |
numbax-x.y.z.n+bu.v.w.m-cp310-cp310-linux_86_64.whl |
适用于Python 3.10的numbax Python二进制扩展包 |
Python wheel包 |
rmmx-x.y.z.n+bu.v.w.m-cp38-cp38-linux_86_64.whl |
适用于Python 3.8的rmmx Python二进制扩展包 |
Python wheel包 |
rmmx-x.y.z.n+bu.v.w.m-cp310-cp310-linux_86_64.whl |
适用于Python 3.10的rmmx Python二进制扩展包 |
Python wheel包 |
dask_mcdf-x.y.z.n+bu.v.w.m-py3-none-any.whl |
适用于Python 3.8或3.10的Dask-mcDF Python二进制扩展包 |
Python wheel包 |
dask_maca-x.y.z.n+bu.v.w.m-py3-none-any.whl |
适用于Python 3.8或3.10的Dask-MACA Python二进制扩展包 |
deb二进制发布包 |
mcdf-x.y.z.n+bu.v.w.m-linux.x86_64.deb |
适用于Ubuntu等使用deb包管理的系统 |
rpm二进制发布包 |
mcdf-x.y.z.n+bu.v.w.m-linux.x86_64.rpm |
适用于CentOS等使用rpm包管理的系统 |
备注
x.y.z.n表示对应包的软件发布版本,bu.v.w.m表示基于版本号为u.v.w.m的MXMACA进行编译的构建号。
mcDF运行中依赖mcPy、numbax和rmmx。
当前支持x86_64平台的Python 3.8和3.10,请选取适配具体Python版本的软件包。
1.2.2. 软件包内容
mcDF C++二进制软件包将会在/opt/mxmap路径下安装相关的二进制开发库及头文件,以/opt/mxmap为起始路径,可以得到如下的文件内容:
.
├── include
│ ├── cudf
│ │ ├── aggregation.hpp
│ │ ├── ast
│ │ ├── ......
│ │ └── unary.hpp
│ ├── cudf_jni
│ │ ├── ColumnViewJni.hpp
│ │ └── ......
│ └── ......
└── lib
├── cmake
│ └── cudf
│ ├── cudf-config.cmake
│ ├── cudf-targets.cmake
│ └── ......
├── libcudf.a
├── libcudf.so
└── ......
include/cudf/目录包含了mcDF所有的头文件,include/cudf_jni是mcDF的JNI头文件
lib/libcudf.a文件是静态库,lib/libcudf.so文件是动态链接库
lib/cmake/cudf目录中的cudf-config.cmake和cudf-targets.cmake等是使用CMake配置mcDF的配置文件,在CMakelists.txt中可以直接使用
find_package引入mcDF动态库以及头文件
2. 安装部署
2.1. 依赖关系
安装mcDF之前需要满足如下硬软件依赖:
曦云系列GPU:mcDF仅支持基于曦云系列芯片的硬件平台
MXMACA SDK:mcDF基于MXMACA SDK实现GPU计算加速,安装mcDF之前必须先安装MXMACA SDK,配套版本以《MXMAP发布说明》为准
mcDF依赖基于MXMACA SDK的numbax、rmmx、mcPy等Python包
Dask-mcDF的本地分布式功能依赖基于MXMACA SDK的Dask-MACA Python包
Ubuntu 18.04 LTS、Ubuntu 20.04 LTS、Ubuntu 22.04 LTS、CentOS 8,且gcc/g++ 9.5+
x86_64,glibc≥2.27
Python 3.8或3.10
2.2. mcDF安装与卸载
安装mcDF之前需要安装沐曦编译发布的numbax、rmmx、mcPy Python包。
备注
mcDF、mcPy、numbax等依赖的第三方Python包,可以从公开pip源下载安装,本文中将仅描述对第三方Python包的依赖关系,不描述具体的安装指导。
如果mcDF的目标安装环境支持从pip源在线下载安装Python包,则在mcDF、mcPy、numbax等包的安装过程中会自动下载安装第三方依赖。
如果mcDF的目标安装环境不支持从pip源在线下载安装Python包,则用户需将第三方依赖库离线下载到本地,然后执行pip install命令进行安装。
2.2.1. 安装mcDF
numbax、rmmx、mcPy之间存在依赖关系,需按照文档中描述的先后顺序安装。
安装numbax。
依赖说明:
zipp>=0.5 cython llvmlite>=0.39.0dev0,<0.40 Numpy>=1.21,<=1.25 setuptools>=65.5.1 importlib_metadata
将numbax wheel包下载到本地,执行以下命令进行安装,安装过程中会自动尝试从pip源下载安装上述依赖库。
pip install numbax-x.y.z.n+bu.v.w.m-cp38-cp38-linux_x86-64.whl
或
pip install numbax-x.y.z.n+bu.v.w.m-cp310-cp310-linux_x86-64.whl
安装rmmx。
依赖说明:
Numpy>=1.19
将rmmx wheel包下载到本地,执行以下命令进行安装,安装过程中会自动尝试从pip源下载安装上述依赖库。
pip install rmmx-x.y.z.n+bu.v.w.m-cp38-cp38-linux_x86-64.whl
或
pip install rmmx-x.y.z.n+bu.v.w.m-cp310-cp310-linux_x86-64.whl
安装mcPy。
依赖说明:
Numpy>=1.21,<=1.27 FastRLock>=0.5 numbax>=0.56
将mcPy wheel包下载到本地,执行以下命令进行安装,安装过程中会自动尝试从pip源下载安装上述依赖库。
pip install mcpy-x.y.z.n+bu.v.w.m-cp38-cp38-linux_x86-64.whl
或
pip install mcpy-x.y.z.n+bu.v.w.m-cp310-cp310-linux_x86-64.whl
安装mcDF。
依赖说明:
cachetools fsspec>=0.6.0 packaging pandas>=1.0,<1.6.0dev0 protobuf>=3.20.1,<3.21.0a0 typing_extensions pyarrow==9.0.0
将mcDF wheel包下载到本地,执行以下命令进行安装,安装过程中会自动尝试从pip源下载安装上述依赖库。
pip install mcdf-x.y.z.n+bu.v.w.m-cp38-cp38-linux_x86-64.whl
或
pip install mcdf-x.y.z.n+bu.v.w.m-cp310-cp310-linux_x86-64.whl
2.2.2. 安装Dask-mcDF
使用Dask-mcDF的本地分布式功能需要安装Dask-MACA Python包。
安装Dask-MACA。
依赖说明:
dask==2023.1.1 distributed==2023.1.1 numpy>=1.18.0 pandas>=1.0 zict>=0.1.3
将Dask-MACA wheel包下载到本地,并执行以下命令进行安装,安装过程中会自动尝试从pip源下载安装上述依赖库。
pip install dask_maca-x.y.z.n+bu.v.w.m-py3-none-any.whl
安装Dask-mcDF。
依赖说明:
dask==2023.1.1 distributed>=2023.1.1 fsspec>=0.6.0 numpy panda>=1.0,<1.6.0dev0 mcdf mcpy
将Dask-mcDF wheel包下载到本地,并执行以下命令进行安装,安装过程中会自动尝试从pip源下载安装上述依赖库。
pip install dask_mcdf-x.y.z.n+bu.v.w.m-py3-none-any.whl
2.2.3. 安装mcDF(C++)
安装mcDF(C++)前需要手动卸载之前安装的mcDF的deb或者rpm包。
安装依赖
安装GCC
由于mcDF基于C++17新特性开发,需要GCC 9.5及以上版本,libstdc++(版本高于libstdc++.so.6.0.28),对于Ubuntu 18.04以及CentOS 8需要手动安装GCC到 /usr/ 目录。
配置Conda依赖
mcDF还需要libarrow等第三方库。推荐使用Conda新建环境,并安装以下依赖项。注意,使用libarrow而不要使用pyarrow。
- aiobotocore>=2.2.0 - boto3>=1.21.21 - botocore>=1.24.21 - c-compiler - cachetools - cmake>=3.23.1,<3.25.0 - cxx-compiler - dask>=2022.9.2 - distributed>=2022.9.2 - dlpack>=0.5,<0.6.0a0 - doxygen=1.8.20 - fastavro>=0.22.9 - fsspec>=0.6.0 - gcc_linux-64=9.* - hypothesis - libarrow=10 - librdkafka=1.7.0 - mimesis>=4.1.0 - moto>=4.0.8 - myst-nb - nbsphinx - packaging - pip - pre-commit - protobuf=4.21 - s3fs>=2022.3.0 - streamz - sysroot_linux-64==2.17 - typing_extensions - spdlog=1.8.5 - fastrlock>=0.5 - llvmlite>=0.39.0dev0,<0.40 - xsimd=8.1.0 - bcrypt=4.0.1
安装mcDF(C++)
Ubuntu系统
假设mcDF(C++)的deb包已经保存在当前目录下,则可以使用
dpkg安装:$ sudo dpkg -i ./mcdf-x.y.z.n+bu.v.w.m-linux.x86_64.deb
安装成功后可以在 /opt/mxmap下找到mcDF对应的头文件,动态库,以及CMake文件。也可以通过以下命令检查是否安装成功:
$ dpkg -l | grep mcdf
CentOS系统
假设mcDF(C++)的rpm包已经保存在当前目录,则可以使用如下命令安装。其中
prefix为MXMACA u.v.w.m的真实路径:$ sudo rpm -ivh ./mcdf-x.y.z.n+bu.v.w.m-linux.x86_64.rpm
安装成功后可以在 /opt/mxmap下找到mcDF对应的头文件,动态库,以及CMake文件。也可以通过以下命令检查是否安装成功:
$ rpm -qa | grep mcdf
备注
u.v.w.m为MXMACA的发布版本。
2.2.4. 卸载
执行以下命令,卸载mcDF。
$ pip uninstall mcdf # python package
$ pip uninstall dask_mcdf # python package
$ sudo dpkg -r mcdf # deb package
$ sudo rpm -e mcdf # rpm package
2.3. 环境变量设置
mcDF正常运行依赖如下环境变量:
MACA_PATH:指向MXMACA包的安装路径
MXMACA安装和运行所需要的其他环境变量
如果应用程序使用的GPU设备的数量少于实际数量,为了获得最佳性能,建议通过 MACA_VISIBLE_DEVICES 环境变量指定实际使用的GPU ID。
2.4. 常见问题
2.4.1. case1:numbax报错
如果遇到numbax报错如下:
"initialization of _internal failed without raising an exception"
请检查NumPy版本,推荐1.21 ≤ NumPy ≤ 1.25。
2.4.2. case2:关于pyarrow版本依赖关系
当采用mcDF wheel包安装部署方式时,会在mcDF的安装路径下同时安装libcudf.so和libarrow.so,这两个so之间存在配套绑定关系。
如果运行环境为conda虚拟环境,并安装了arrow-cpp和pyarrow,此时运行环境中将存在两个libarrow.so,一个是arrow-cpp中安装的,另一个是mcDF中安装的。
如果在Python文件执行过程中先加载arrow-cpp的libarrow.so,那么后续执行 import cudf 时将会遇到libarrow.so中符号缺失的问题。
解决方法:
conda虚拟环境中同时安装arrow-cpp、pyarrow与仅pip安装pyarrow,这两种情况下安装的libarrow.so存在非常大的不同。
一般情况下,不会在运行时的conda虚拟环境中同时安装arrow-cpp和pyarrow。但如果同时安装了arrow-cpp和pyarrow,推荐采用mcDF的镜像发布方式。
如果可以修改运行时的conda虚拟环境,可以仅安装pyarrow而不安装arrow-cpp。
2.4.3. case3:关于glibc版本配套
mcDF运行时的glibc版本需≥glibc 2.27,否则可能存在符号缺失的问题。
可通过 ldd –version 命令查询当前环境中的glibc版本。
3. mcDF用户指南
3.1. mcDF和Dask-mcDF
mcDF是一个Python GPU DataFrame库(基于Apache Arrow列式内存格式构建),用于以一种类似于pandas的方式来加载、连接、聚合、筛选、操作DataFrame样式的表格数据。
Dask是一个灵活的Python并行计算库,能够平滑而简单地扩展工作流程。在CPU上,Dask使用pandas在DataFrame分区上并行执行操作。
Dask-mcDF在Dask基础上进行了必要扩展,以允许使用mcDF GPU DataFrame而不是pandas DataFrame处理其DataFrame分区。
例如,当调用 dask_cudf.read_csv(…) 时,集群的GPU通过调用 cudf.read_csv 来解析csv文件而不是调用 pandas.read_csv 来解析csv文件。
3.1.1. mcDF和Dask-mcDF使用场景
如果工作流程在单个GPU上足够快,或者数据可以轻松地放入单个GPU上的内存,那么建议使用mcDF。 如果想将工作流程分布在多个GPU上,拥有超过单个GPU内存容量的数据,或者想一次分析分布在许多文件中的数据,则需要使用Dask-mcDF。
import os
import cupy as cp
import pandas as pd
import cudf
import dask_cudf
cp.random.seed(12)
#### Portions of this were borrowed and adapted from the
#### mcDF cheatsheet, existing mcDF documentation,
#### and 10 Minutes to pandas.
3.1.2. 创建对象
创建一个 cudf.Series 对象和一个 dask_cudf.Series 对象:
>>> s = cudf.Series([1, 2, 3, None, 4])
>>> s
0 1
1 2
2 3
3 <NA>
4 4
dtype: int64
>>> ds = dask_cudf.from_cudf(s, npartitions=2)
# Note the call to head here to show the first few entries.
>>> ds.head(n=3)
0 1
1 2
2 3
dtype: int64
通过为每列指定初始值来创建一个 cudf.Series 对象和一个 dask_cudf.Series 对象:
>>> df = cudf.DataFrame(
{
"a": list(range(20)),
"b": list(reversed(range(20))),
"c": list(range(20)),
}
)
>>> df
a b c
0 0 19 0
1 1 18 1
2 2 17 2
3 3 16 3
4 4 15 4
5 5 14 5
6 6 13 6
7 7 12 7
8 8 11 8
9 9 10 9
10 10 9 10
11 11 8 11
12 12 7 12
13 13 6 13
14 14 5 14
15 15 4 15
16 16 3 16
17 17 2 17
18 18 1 18
19 19 0 19
把mcDF数据帧转换为一个Dask-mcDF数据帧:
两者的关键区别在于:如果要检查数据,在mcDF中可以直接打印出DataFrame对象的值,而在Dask-mcDF中必须调用 .head() 来查看前几个值。一般情况下,ddf中的数据将分布在多个GPU中。
在这种小数据量的情况下,可以调用 ddf.compute() 从Dask-mcDF对象中获取一个mcDF对象。
一般来说,应该避免在大型数据帧上调用 .compute(),同时也应限制在一些相对小的后处理结果上使用它。
因此,在本文档中,通常会调用 .head() 来检查Dask-mcDF数据帧的前几个值,偶尔会调用 .compute()。
>>> ddf = dask_cudf.from_cudf(df, npartitions=2)
>>> ddf.head()
a b c
0 0 19 0
1 1 18 1
2 2 17 2
3 3 16 3
4 4 15 4
从pandas数据帧创建一个mcDF数据帧,以及从mcDF数据帧创建一个Dask-mcDF数据帧:
请注意,使用Dask-mcDF的最佳实践是使用 read_csv 或其他内置I/O例程(如下所述)将数据直接读取到 dask_cudf.DataFrame 中。
>>> pdf = pd.DataFrame({"a": [0, 1, 2, 3], "b": [0.1, 0.2, None, 0.3]})
>>> gdf = cudf.DataFrame.from_pandas(pdf)
>>> gdf
a b
0 0 0.1
1 1 0.2
2 2 <NA>
3 3 0.3
>>> dask_gdf = dask_cudf.from_cudf(gdf, npartitions=2)
>>> dask_gdf.head(n=2)
a b
0 0 0.1
1 1 0.2
3.1.3. 查看数据
查看GPU数据帧的前几行:
>>> df.head(2)
a b c
0 0 19 0
1 1 18 1
>>> ddf.head(2)
a b c
0 0 19 0
1 1 18 1
根据值大小进行排序:
>>> df.sort_values(by="b")
a b c
19 19 0 19
18 18 1 18
17 17 2 17
16 16 3 16
15 15 4 15
14 14 5 14
13 13 6 13
12 12 7 12
11 11 8 11
10 10 9 10
9 9 10 9
8 8 11 8
7 7 12 7
6 6 13 6
5 5 14 5
4 4 15 4
3 3 16 3
2 2 17 2
1 1 18 1
0 0 19 0
>>> ddf.sort_values(by="b").head()
a b c
19 19 0 19
18 18 1 18
17 17 2 17
16 16 3 16
15 15 4 15
3.1.4. 选择数据
3.1.4.1. 选择列
选择一个单独的列:
>>> df["a"]
0 0
1 1
2 2
3 3
4 4
5 5
6 6
7 7
8 8
9 9
10 10
11 11
12 12
13 13
14 14
15 15
16 16
17 17
18 18
19 19
Name: a, dtype: int64
>>> ddf["a"].head()
0 0
1 1
2 2
3 3
4 4
Name: a, dtype: int64
3.1.4.2. 根据标签选择
选择a列和b列的第2到第5行:
>>> df.loc[2:5, ["a", "b"]]
a b
2 2 17
3 3 16
4 4 15
5 5 14
>>> ddf.loc[2:5, ["a", "b"]].head()
a b
2 2 17
3 3 16
4 4 15
5 5 14
3.1.4.3. 根据位置索引选择
和NumPy和pandas一样,通过一个或者多个整数位置索引切片的方式选择数据:
请注意这种方式不适用于Dask-mcDF DataFrames。
>>> df.iloc[0]
a 0
b 19
c 0
Name: 0, dtype: int64
>>> df.iloc[0:3, 0:2]
a b
0 0 19
1 1 18
2 2 17
也可以直接通过索引的方式选择DataFrame或者Series中的元素:
>>> df[3:5]
a b c
3 3 16 3
4 4 15 4
>>> s[3:5]
3 <NA>
4 4
dtype: int64
3.1.5. 布尔索引
通过布尔索引在DataFrame或者Series中选择行数据:
>>> df[df.b > 15]
a b c
0 0 19 0
1 1 18 1
2 2 17 2
3 3 16 3
>>> ddf[ddf.b > 15].head(n=3)
a b c
0 0 19 0
1 1 18 1
2 2 17 2
通过query API选择行数据:
>>> df.query("b == 3")
a b c
16 16 3 16
请注意,在Dask-mcDF数据帧上调用 compute() 而不是 head(),因为确认匹配的行数会很小,因此,将整个结果返回是合理的。
>>> ddf.query("b == 3").compute()
a b c
16 16 3 16
还可以通过 local_dict 关键字将局部变量传递给Dask-mcDF查询。对于标准的mcDF,可以使用 local_dict 关键字,也可以通过 @ 关键字直接传递变量。
支持的逻辑运算符包括>、<、>=、<=、==、和!=。
>>> cudf_comparator = 3
>>> df.query("b == @cudf_comparator")
a b c
16 16 3 16
>>> dask_cudf_comparator = 3
>>> ddf.query("b == @val", local_dict={"val": dask_cudf_comparator}).compute()
a b c
16 16 3 16
使用 isin 方法来查询数据:
>>> df[df.a.isin([0, 5])]
a b c
0 0 19 0
5 5 14 5
3.1.6. 多重索引(MultiIndex)
mcDF支持使用多重索引对数据帧进行分层索引。分组会自动生成一个具有多索引的数据帧,参见 3.1.8.7 分组(Grouping)。
arrays = [["a", "a", "b", "b"], [1, 2, 3, 4]]
tuples = list(zip(*arrays))
idx = cudf.MultiIndex.from_tuples(tuples)
idx
MultiIndex([('a', 1),
('a', 2),
('b', 3),
('b', 4)],
)
此索引可以用于数据帧的Index轴或者Column轴:
>>> gdf1 = cudf.DataFrame(
{"first": cp.random.rand(4), "second": cp.random.rand(4)}
)
>>> gdf1.index = idx
>>> gdf1
first second
a 1 0.082654 0.967955
2 0.399417 0.441425
b 3 0.784297 0.793582
4 0.070303 0.271711
>>> gdf2 = cudf.DataFrame(
{"first": cp.random.rand(4), "second": cp.random.rand(4)}
).T
>>> gdf2.columns = idx
>>> gdf2
a b
1 2 3 4
First 0.343382 0.003700 0.20043 0.581614
second 0.907812 0.101512 0.24179 0.224180
使用多重索引访问数据帧的值,使用 .loc :
>>> gdf1.loc[("b", 3)]
first 0.784297
second 0.793582
Name: ('b', 3), dtype: float64
以及 .iloc :
>>> gdf1.iloc[0:2]
first second
a 1 0.082654 0.967955
2 0.399417 0.441425
3.1.7. 缺失数据处理
可以使用 fillna 方法替换缺失的数据:
>>> s.fillna(999)
0 1
1 2
2 3
3 999
4 4
dtype: int64
>>> ds.fillna(999).head(n=3)
0 1
1 2
2 3
dtype: int64
3.1.8. 数据操作方法
3.1.8.1. 统计
计算Series的描述性统计信息:
>>> s.mean(), s.var()
(2.5, 1.666666666666666)
下面是一个典型的例子,说明什么时候可以调用 .compute()。在任何情况下,平均值和方差的计算结果都是一个数字,所以使用 .compute() 而不是使用 .head() 查看结果是合理的。
>>> ds.mean().compute(), ds.var().compute()
(2.5, 1.6666666666666667)
3.1.8.2. 应用转换函数(Applymap)
将函数应用于Series。请注意,直接使用Dask-mcDF应用用户定义的函数尚未实现。目前,可以使用 map_partitions 将一个函数应用于分布式数据帧的每个分区。
def add_ten(num):
return num + 10
df["a"].apply(add_ten)
0 10
1 11
2 12
3 13
4 14
5 15
6 16
7 17
8 18
9 19
10 20
11 21
12 22
13 23
14 24
15 25
16 26
17 27
18 28
19 29
Name: a, dtype: int64
>>> ddf["a"].map_partitions(add_ten).head()
0 10
1 11
2 12
3 13
4 14
Name: a, dtype: int64
3.1.8.3. 计算直方图(Histogramming)
计算每一个唯一值变量的出现次数:
>>> df.a.value_counts()
15 1
6 1
1 1
14 1
2 1
5 1
11 1
7 1
17 1
13 1
8 1
16 1
0 1
10 1
4 1
9 1
19 1
18 1
3 1
12 1
Name: a, dtype: int32
>>> ddf.a.value_counts().head()
15 1
6 1
1 1
14 1
2 1
Name: a, dtype: int64
3.1.8.4. 字符串方法
与pandas一样,mcDF在Series的 str 属性中提供了字符串处理方法:
>>> s = cudf.Series(["A", "B", "C", "Aaba", "Baca", None, "CABA", "dog", "cat"])
>>> s.str.lower()
0 a
1 b
2 c
3 aaba
4 baca
5 <NA>
6 caba
7 dog
8 cat
dtype: object
>>> ds = dask_cudf.from_cudf(s, npartitions=2)
>>> ds.str.lower().head(n=4)
0 a
1 b
2 c
3 aaba
dtype: object
除了简单的操作外,还可以使用正则表达式匹配字符串:
>>> s.str.match("^[aAc].+")
0 False
1 False
2 False
3 True
4 False
5 <NA>
6 False
7 False
8 True
dtype: bool
>>> ds.str.match("^[aAc].+").head()
0 False
1 False
2 False
3 True
4 False
dtype: bool
3.1.8.5. 连接(Concat)
按行连接Series和DataFrame:
>>> s = cudf.Series([1, 2, 3, None, 5])
>>> cudf.concat([s, s])
0 1
1 2
2 3
3 <NA>
4 5
0 1
1 2
2 3
3 <NA>
4 5
dtype: int64
>>> ds2 = dask_cudf.from_cudf(s, npartitions=2)
>>> dask_cudf.concat([ds2, ds2]).head(n=3)
0 1
1 2
2 3
dtype: int64
3.1.8.6. 合并(Join)
执行SQL样式的合并。请注意,数据帧的顺序不会得到保持,但可以在合并后通过按索引排序恢复。
>>> df_a = cudf.DataFrame()
>>> df_a["key"] = ["a", "b", "c", "d", "e"]
>>> df_a["vals_a"] = [float(i + 10) for i in range(5)]
>>> df_b = cudf.DataFrame()
>>> df_b["key"] = ["a", "c", "e"]
>>> df_b["vals_b"] = [float(i + 100) for i in range(3)]
>>> merged = df_a.merge(df_b, on=["key"], how="left")
>>> merged
key vals_a vals_b
0 a 10.0 100.0
1 c 12.0 101.0
2 e 14.0 102.0
3 b 11.0 <NA>
4 d 13.0 <NA>
>>> ddf_a = dask_cudf.from_cudf(df_a, npartitions=2)
>>> ddf_b = dask_cudf.from_cudf(df_b, npartitions=2)
>>> merged = ddf_a.merge(ddf_b, on=["key"], how="left").head(n=4)
>>> merged
key vals_a vals_b
0 c 12.0 101.0
1 e 14.0 102.0
2 b 11.0 <NA>
3 d 13.0 <NA>
3.1.8.7. 分组(Grouping)
与pandas一样,mcDF和Dask-mcDF支持按组Split-Apply-Combine模式:
df["agg_col1"] = [1 if x % 2 == 0 else 0 for x in range(len(df))]
df["agg_col2"] = [1 if x % 3 == 0 else 0 for x in range(len(df))]
ddf = dask_cudf.from_cudf(df, npartitions=2)
分组,然后对分组后的数据应用求和函数:
>>> df.groupby("agg_col1").sum()
a b c agg_col2
agg_col1
1 90 100 90 4
0 100 90 100 3
>>> ddf.groupby("agg_col1").sum().compute()
a b c agg_col2
agg_col1
1 90 100 90 4
0 100 90 100 3
分组,然后对分组数据应用求和函数:
>>> df.groupby(["agg_col1", "agg_col2"]).sum()
a b c
agg_col1 agg_col2
1 0 54 60 54
0 0 73 60 73
1 1 36 40 36
0 1 27 30 27
>>> ddf.groupby(["agg_col1", "agg_col2"]).sum().compute()
a b c
agg_col1 agg_col2
1 1 36 40 36
0 0 73 60 73
1 0 54 60 54
0 1 27 30 27
使用 agg 对特定列进行分组并应用统计函数:
>>> df.groupby("agg_col1").agg({"a": "max", "b": "mean", "c": "sum"})
a b c
agg_col1
1 18 10.0 90
0 19 9.0 100
>>> ddf.groupby("agg_col1").agg({"a": "max", "b": "mean", "c": "sum"}).compute()
a b c
agg_col1
1 18 10.0 90
0 19 9.0 100
3.1.8.8. 数据转置(Transpose)
使用 transpose 方法或T属性来转置DataFrame,当前的实现要求所有列必须具有相同的数据类型。目前在Dask-mcDF中未实施转置功能。
>>> sample = cudf.DataFrame({"a": [1, 2, 3], "b": [4, 5, 6]})
>>> sample
a b
0 1 4
1 2 5
2 3 6
>>> sample.transpose()
0 1 2
a 1 2 3
b 4 5 6
3.1.8.9. 时间序列(Time Series)
DataFrame支持日期时间类型的列,允许用户根据特定的时间戳与数据交互并筛选数据。
>>> import datetime as dt
>>> date_df = cudf.DataFrame()
>>> date_df["date"] = pd.date_range("11/20/2018", periods=72, freq="D")
>>> date_df["value"] = cp.random.sample(len(date_df))
>>> search_date = dt.datetime.strptime("2018-11-23", "%Y-%m-%d")
>>> date_df.query("date <= @search_date")
date value
0 2018-11-20 0.986051
1 2018-11-21 0.232034
2 2018-11-22 0.397617
3 2018-11-23 0.103839
>>> date_ddf = dask_cudf.from_cudf(date_df, npartitions=2)
>>> date_ddf.query(
"date <= @search_date", local_dict={"search_date": search_date}
).compute()
date value
0 2018-11-20 0.986051
1 2018-11-21 0.232034
2 2018-11-22 0.397617
3 2018-11-23 0.103839
3.1.8.10. 分类(Categoricals)
DataFrame支持分类列:
>>> gdf = cudf.DataFrame(
{"id": [1, 2, 3, 4, 5, 6], "grade": ["a", "b", "b", "a", "a", "e"]}
)
>>> gdf["grade"] = gdf["grade"].astype("category")
>>> gdf
id grade
0 1 a
1 2 b
2 3 b
3 4 a
4 5 a
5 6 e
>>> dgdf = dask_cudf.from_cudf(gdf, npartitions=2)
>>> dgdf.head(n=3)
id grade
0 1 a
1 2 b
2 3 b
访问列的类别。请注意,Dask-mcDF目前不支持此功能。
gdf.grade.cat.categories
StringIndex(['a' 'b' 'e'], dtype='object')
访问每个分类观察的基本代码值:
>>> gdf.grade.cat.codes
0 0
1 1
2 1
3 0
4 0
5 2
dtype: uint8
>>> dgdf.grade.cat.codes.compute()
0 0
1 1
2 1
3 0
4 0
5 2
dtype: uint8
3.1.9. 数据转换
3.1.9.1. pandas
将mcDF和Dask-mcDF数据帧转换为pandas数据帧:
>>> df.head().to_pandas()
a b c agg_col1 agg_col2
0 0 19 0 1 1
1 1 18 1 0 0
2 2 17 2 1 0
3 3 16 3 0 1
4 4 15 4 1 0
为了将前几个条目转换为pandas,类似地,在Dask-mcDF数据帧上调用 .head() 来获得本地mcDF数据框架,然后可以对其进行转换。
>>> ddf.head().to_pandas()
a b c agg_col1 agg_col2
0 0 19 0 1 1
1 1 18 1 0 0
2 2 17 2 1 0
3 3 16 3 0 1
4 4 15 4 1 0
相比之下,如果想转换整个帧,需要在ddf上调用 .compute() 来获得本地mcDF数据帧,然后调用 to_pandas(),再进行后续处理。不太推荐使用此工作流,因为它既给单个GPU带来了很高的内存压力( .compute() 调用),又没有利用GPU加速进行处理(在pandas中进行计算)。
>>> ddf.compute().to_pandas().head()
a b c agg_col1 agg_col2
0 0 19 0 1 1
1 1 18 1 0 0
2 2 17 2 1 0
3 3 16 3 0 1
4 4 15 4 1 0
3.1.9.2. NumPy
将mcDF和Dask-mcDF数据帧转换为NumPy ndarray:
>>> df.to_numpy()
array([[ 0, 19, 0, 1, 1],
[ 1, 18, 1, 0, 0],
[ 2, 17, 2, 1, 0],
[ 3, 16, 3, 0, 1],
[ 4, 15, 4, 1, 0],
[ 5, 14, 5, 0, 0],
[ 6, 13, 6, 1, 1],
[ 7, 12, 7, 0, 0],
[ 8, 11, 8, 1, 0],
[ 9, 10, 9, 0, 1],
[10, 9, 10, 1, 0],
[11, 8, 11, 0, 0],
[12, 7, 12, 1, 1],
[13, 6, 13, 0, 0],
[14, 5, 14, 1, 0],
[15, 4, 15, 0, 1],
[16, 3, 16, 1, 0],
[17, 2, 17, 0, 0],
[18, 1, 18, 1, 1],
[19, 0, 19, 0, 0]])
>>> ddf.compute().to_numpy()
array([[ 0, 19, 0, 1, 1],
[ 1, 18, 1, 0, 0],
[ 2, 17, 2, 1, 0],
[ 3, 16, 3, 0, 1],
[ 4, 15, 4, 1, 0],
[ 5, 14, 5, 0, 0],
[ 6, 13, 6, 1, 1],
[ 7, 12, 7, 0, 0],
[ 8, 11, 8, 1, 0],
[ 9, 10, 9, 0, 1],
[10, 9, 10, 1, 0],
[11, 8, 11, 0, 0],
[12, 7, 12, 1, 1],
[13, 6, 13, 0, 0],
[14, 5, 14, 1, 0],
[15, 4, 15, 0, 1],
[16, 3, 16, 1, 0],
[17, 2, 17, 0, 0],
[18, 1, 18, 1, 1],
[19, 0, 19, 0, 0]])
将mcDF或者Dask-mcDF Series转换为NumPy ndarray:
>>> df["a"].to_numpy()
array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19])
>>> ddf["a"].compute().to_numpy()
array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19])
3.1.9.3. Arrow
将mcDF和Dask-mcDF数据帧转换为PyArrow Table:
>>> df.to_arrow()
pyarrow.Table
a: int64
b: int64
c: int64
agg_col1: int64
agg_col2: int64
----
a: [[0,1,2,3,4,...,15,16,17,18,19]]
b: [[19,18,17,16,15,...,4,3,2,1,0]]
c: [[0,1,2,3,4,...,15,16,17,18,19]]
agg_col1: [[1,0,1,0,1,...,0,1,0,1,0]]
agg_col2: [[1,0,0,1,0,...,1,0,0,1,0]]
>>> ddf.head().to_arrow()
pyarrow.Table
a: int64
b: int64
c: int64
agg_col1: int64
agg_col2: int64
----
a: [[0,1,2,3,4]]
b: [[19,18,17,16,15]]
c: [[0,1,2,3,4]]
agg_col1: [[1,0,1,0,1]]
agg_col2: [[1,0,0,1,0]]
3.1.10. I/O操作
3.1.10.1. CSV
将数据写入一个CSV文件:
if not os.path.exists("example_output"):
os.mkdir("example_output")
df.to_csv("example_output/foo.csv", index=False)
ddf.compute().to_csv("example_output/foo_dask.csv", index=False)
从CSV文件中读取数据:
>>> df = cudf.read_csv("example_output/foo.csv")
>>> df
a b c agg_col1 agg_col2
0 0 19 0 1 1
1 1 18 1 0 0
2 2 17 2 1 0
3 3 16 3 0 1
4 4 15 4 1 0
5 5 14 5 0 0
6 6 13 6 1 1
7 7 12 7 0 0
8 8 11 8 1 0
9 9 10 9 0 1
10 10 9 10 1 0
11 11 8 11 0 0
12 12 7 12 1 1
13 13 6 13 0 0
14 14 5 14 1 0
15 15 4 15 0 1
16 16 3 16 1 0
17 17 2 17 0 0
18 18 1 18 1 1
19 19 0 19 0 0
对于Dask-mcDF,优先使用 dask_cudf.read_csv,而不是 dask_cudf.from_cudf(cudf.read_csv),因为前者可以跨多个GPU并行化,并处理更大的CSV文件。
>>> ddf = dask_cudf.read_csv("example_output/foo_dask.csv")
>>> ddf.head()
a b c agg_col1 agg_col2
0 0 19 0 1 1
1 1 18 1 0 0
2 2 17 2 1 0
3 3 16 3 0 1
4 4 15 4 1 0
使用星号通配符将目录中的所有CSV文件读取到单个dask_cudf.DataFrame中:
>>> ddf = dask_cudf.read_csv("example_output/*.csv")
>>> ddf.head()
a b c agg_col1 agg_col2
0 0 19 0 1 1
1 1 18 1 0 0
2 2 17 2 1 0
3 3 16 3 0 1
4 4 15 4 1 0
3.1.10.2. Parquet
写入parquet文件:
df.to_parquet("example_output/temp_parquet")
读取parquet文件:
>>> df = cudf.read_parquet("example_output/temp_parquet")
>>> df
a b c agg_col1 agg_col2
0 0 19 0 1 1
1 1 18 1 0 0
2 2 17 2 1 0
3 3 16 3 0 1
4 4 15 4 1 0
5 5 14 5 0 0
6 6 13 6 1 1
7 7 12 7 0 0
8 8 11 8 1 0
9 9 10 9 0 1
10 10 9 10 1 0
11 11 8 11 0 0
12 12 7 12 1 1
13 13 6 13 0 0
14 14 5 14 1 0
15 15 4 15 0 1
16 16 3 16 1 0
17 17 2 17 0 0
18 18 1 18 1 1
19 19 0 19 0 0
将dask_cudf.DataFrame写入parquet文件:
ddf.to_parquet("example_output/ddf_parquet_files")
3.1.10.3. ORC
写入ORC文件:
df.to_orc("example_output/temp_orc")
读取ORC文件:
>>> df2 = cudf.read_orc("example_output/temp_orc")
>>> df2
a b c agg_col1 agg_col2
0 0 19 0 1 1
1 1 18 1 0 0
2 2 17 2 1 0
3 3 16 3 0 1
4 4 15 4 1 0
5 5 14 5 0 0
6 6 13 6 1 1
7 7 12 7 0 0
8 8 11 8 1 0
9 9 10 9 0 1
10 10 9 10 1 0
11 11 8 11 0 0
12 12 7 12 1 1
13 13 6 13 0 0
14 14 5 14 1 0
15 15 4 15 0 1
16 16 3 16 1 0
17 17 2 17 0 0
18 18 1 18 1 1
19 19 0 19 0 0
3.2. mcDF与pandas的比较
mcDF是一个 处理DataFrame的库,与pandas API非常类似,但它不是pandas的完全替代品。在API和行为方面,mcDF和pandas之间存在一些差异。
3.2.1. 支持的操作方法
mcDF支持许多与pandas相同的数据结构和操作。这包括Series、DataFrame、Index和对它们的操作,如一元和二元操作、索引、筛选、连接、分组和窗口操作等。
3.2.2. 数据类型
mcDF支持pandas中许多常用的数据类型,包括numeric、datetime、timestamp、string和categorical数据类型。此外,支持一些特殊的数据类型例如decimal、list和 “struct”。 有关详细信息,参见3.3 支持的数据类型。
备注
不支持像pandas的ExtensionDtype这样的自定义数据类型。
3.2.3. Null或缺失数据
与pandas不同,mcDF中的所有数据类型都可以为null,这意味着它们可能包含缺失的值(由 cudf.NA 表示)。
>>> s = cudf.Series([1, 2, cudf.NA])
>>> s
0 1
1 2
2 <NA>
dtype: int64
在任何情况下,Null都不会被强制为NaN;将mcDF的行为与pandas的行为进行比较如下:
>>> s = cudf.Series([1, 2, cudf.NA], dtype="category")
>>> s
0 1
1 2
2 <NA>
dtype: category
Categories (2, int64): [1, 2]
>>> s = pd.Series([1, 2, pd.NA], dtype="category")
>>> s
0 1
1 2
2 NaN
dtype: category
Categories (2, int64): [1, 2]
3.2.4. 迭代
不支持在mcDF Series、DataFrame或Index上迭代。这是因为迭代驻留在GPU上的数据会产生极低的性能,因为GPU是针对高度并行操作而不是顺序操作进行优化的。
在绝大多数情况下,可以避免迭代,并使用现有的函数或方法来完成相同的任务。
如果无法避免,请使用 .to_arrow() 或 .to_pandas() 将数据从GPU复制到CPU,然后使用 .from_arrow() 和 .from_pandas() 将结果复制回GPU。
3.2.5. 输出结果排序
默认情况下,mcDF中的 join (或 merge )和 groupby 操作不能保证输出顺序。pandas和mcDF获得的结果比较如下:
>>> import cupy as cp
>>> df = cudf.DataFrame({'a': cp.random.randint(0, 1000, 1000), 'b': range(1000)})
>>> df.groupby("a").mean().head()
b
a
742 694.5
29 840.0
459 525.5
442 363.0
666 7.0
>>> df.to_pandas().groupby("a").mean().head()
b
a
2 643.75
6 48.00
7 631.00
9 906.00
10 640.00
为了和pandas的实现表现一致,必须显式指定sort=True:
>>> df.to_pandas().groupby("a", sort=True).mean().head()
b
a
2 643.75
6 48.00
7 631.00
9 906.00
10 640.00
3.2.6. 浮点运算
mcDF利用GPU并行执行操作。这意味着操作的顺序并不总是确定的。这影响了浮点运算的确定性,因为浮点运算是non-associative,即a+b不等于b+a。
例如,当s是一系列浮点值时, s.sum() 不能保证产生与pandas相同的结果,也不能保证从一次运行到另一次运行产生相同的结果。
如果需要比较浮点结果,通常应该使用cudf.testing模块中提供的函数来进行比较,该模块允许将值比较到所需的精度。
3.2.7. 列名称
与pandas不同,mcDF不支持重复的列名。最好为列名使用唯一的字符串。
3.2.8. “object”数据类型
在pandas和NumPy中, "object" 数据类型用于任意Python对象的集合。例如,在pandas中,可以执行以下操作:
>>> import pandas as pd
>>> s = pd.Series(["a", 1, [1, 2, 3]])
0 a
1 1
2 [1, 2, 3]
dtype: object
为了与pandas兼容,mcDF将字符串的数据类型报告为 "object",但不支持存储或操作任意Python object的集合。
3.2.9. .apply()函数限制
pandas中的 .apply() 函数接受用户定义函数(UDF),该函数可以包括应用于Series、DataFrame的每个值的任意操作,或者在 groupby 的情况下,应用于每个组。mcDF当前版本还不支持 .apply() 函数。
3.3. 支持的数据类型
mcDF支持NumPy和pandas支持的许多数据类型,包括numeric、datetime、timedelta、categorical和string数据类型。还提供了特殊的数据类型,用于处理小数、类似列表和类似字典的数据。
mcDF中的所有数据类型都可以为null,详情参见下表。
Kind of data |
Data type(s) |
|---|---|
Signed integer |
|
Unsigned integer |
|
Floating-point |
|
Datetime |
|
Timedelta (duration) |
|
Category |
|
String |
|
Decimal |
|
List |
|
Struct |
|
3.3.1. NumPy数据类型
将NumPy数据类型用于integer、floating、datetime、timedelta和string数据类型。
因此,就像在NumPy中一样, np.dtype("float32")、 np.float32 和 "float32" 都是指定float32数据类型可接受的方法:
>>> import cudf
>>> s = cudf.Series([1, 2, 3], dtype="float32")
>>> s
0 1.0
1 2.0
2 3.0
dtype: float32
3.3.2. 关于object数据类型
mcDF中与字符串数据关联的数据类型为 "np.object"。
>>> import cudf
>>> s = cudf.Series(["abc", "def", "ghi"])
>>> s.dtype
dtype("object")
这是为了与pandas兼容,但可能会产生误导。在NumPy和pandas中,"object"是由任意Python对象(而不仅仅是字符串)组成的与数据类型相关的数据。
但是,mcDF不支持存储任意Python对象。
3.3.3. 小数(Decimal)数据类型
提供用于处理小数的特殊数据类型,即Decimal32Dtype、Decimal64Dtype和Decimal128Dtype。当需要存储精度高于浮点表示所允许精度的值时,请使用这些数据类型。
mcDF中的小数数据类型基于定点表示。小数数据类型由precision和scale组成。precision表示此数据类型的每个值中的总位数。 例如,与十进制值1.023相关联的precision为4。scale是小数点右边的总位数。与值1.023相关联的scale为3。
每个小数数据类型都与最大precision相关联:
>>> cudf.Decimal32Dtype.MAX_PRECISION
9.0
>>> cudf.Decimal64Dtype.MAX_PRECISION
18.0
>>> cudf.Decimal128Dtype.MAX_PRECISION
38
创建小数Series的一种方法是使用 decimal.Decimal 类型的值。
>>> from decimal import Decimal
>>> s = cudf.Series([Decimal("1.01"), Decimal("4.23"), Decimal("0.5")])
>>> s
0 1.01
1 4.23
2 0.50
dtype: decimal128
>>> s.dtype
Decimal128Dtype(precision=3, scale=2)
结果的数据类型(1.01、4.23、0.50)都可以用至少3的精度和至少2的小数位数表示。然而,值1.234需要至少4的精度和至少3的小数位数,所以就不能使用s的这种数据类型来完全表示:
>>> s[1] = Decimal("1.234") # raises an error
3.3.4. 嵌套数据类型(List和Struct)
ListDtype和StructType是mcDF中用于处理类似列表和类似字典数据的特殊数据类型。这些被称为嵌套数据类型,因为它们能够存储列表的列表、列表的结构或列表的列表的结构等。
可以分别从现有的pandas系列列表和字典中创建列表和结构系列:
>>> psr = pd.Series([{'a': 1, 'b': 2}, {'a': 3, 'b': 4}])
>>> psr
0 {'a': 1, 'b': 2}
1 {'a': 3, 'b': 4}
dtype: object
>>> gsr = cudf.from_pandas(psr)
>>> gsr
0 {'a': 1, 'b': 2}
1 {'a': 3, 'b': 4}
dtype: struct
>>> gsr.dtype
StructDtype({'a': dtype('int64'), 'b': dtype('int64')})
或者使用支持嵌套数据的文件格式从磁盘中读取它们:
>>> pdf = pd.DataFrame({"a": [[1, 2], [3, 4, 5], [6, 7, 8]]})
>>> pdf.to_parquet("lists.pq")
>>> gdf = cudf.read_parquet("lists.pq")
>>> gdf
a
0 [1, 2]
1 [3, 4, 5]
2 [6, 7, 8]
>>> gdf["a"].dtype
ListDtype(int64)
3.4. JSON数据操作
3.4.1. 读取JSON数据
默认情况下,mcDF JSON读取器期望使用面向记录的组织方式的输入数据。
面向记录的JSON数据包括根级别的对象数组,数组中的每个对象对应一行。面向记录的JSON数据以“[”开头,以“]”结尾,并忽略未加引号的空白。
JSON数据的另一个常见变体是“JSON行”,其中JSON对象由换行符( \n )分隔,每个对象对应一行。
>>> j = '''[
{"a": "v1", "b": 12},
{"a": "v2", "b": 7},
{"a": "v3", "b": 5}
]'''
>>> df_records = cudf.read_json(j, engine='cudf')
>>> j = '\n'.join([
... '{"a": "v1", "b": 12}',
... '{"a": "v2", "b": 7}',
... '{"a": "v3", "b": 5}'
... ])
>>> df_lines = cudf.read_json(j, lines=True)
>>> df_lines
a b
0 v1 12
1 2 7
2 v3 5
>>> df_records.equals(df_lines)
True
mcDF JSON读取器还支持JSON对象和数组的任意嵌套组合,这些组合映射到结构和列表数据类型。以下示例演示了用于读取嵌套JSON数据的输入和输出:
# Example with columns types:
# list<int> and struct<k:string>
>>> j = '''[
{"list": [0,1,2], "struct": {"k":"v1"}},
{"list": [3,4,5], "struct": {"k":"v2"}}
]'''
>>> df = cudf.read_json(j, engine='cudf')
>>> df
list struct
0 [0, 1, 2] {'k': 'v1'}
1 [3, 4, 5] {'k': 'v2'}
# Example with columns types:
# list<struct<k:int>> and struct<k:list<int>, m:int>
>>> j = '\n'.join([
... '{"a": [{"k": 0}], "b": {"k": [0, 1], "m": 5}}',
... '{"a": [{"k": 1}, {"k": 2}], "b": {"k": [2, 3], "m": 6}}',
... ])
>>> df = cudf.read_json(j, lines=True)
>>> df
a b
0 [{'k': 0}] {'k': [0, 1], 'm': 5}
1 [{'k': 1}, {'k': 2}] {'k': [2, 3], 'm': 6}
3.4.2. 处理大型和小型JSON行文件
对于基于JSON Lines数据的工作负载,mcDF支持如下用于帮助数据处理的读取器选项:对大文件的字节范围支持,以及对小文件的多源支持。
某些工作流需要处理可能超过GPU内存容量的大型JSON行文件。mcDF中的JSON读取器支持字节范围参数,该参数指定起始字节偏移量和字节大小。读取器解析在字节范围内开始的每个记录,因此字节范围不需要与记录边界对齐。为了避免跳过行或读取重复的行,字节范围应该相邻,示例如下所示:
>>> num_rows = 10
>>> j = '\n'.join([
... '{"id":%s, "distance": %s, "unit": "m/s"}' % x \
... for x in zip(range(num_rows), cupy.random.rand(num_rows))
... ])
>>> chunk_count = 4
>>> chunk_size = len(j) // chunk_count + 1
>>> data = []
>>> for x in range(chunk_count):
... d = cudf.read_json(
... j,
... lines=True,
... byte_range=(chunk_size * x, chunk_size),
... )
... data.append(d)
>>> df = cudf.concat(data)
相比之下,有些工作流需要处理许多小的JSON Lines文件。mcDF中的JSON读取器接受可迭代的数据源,而不是在源之间循环并连接生成的数据帧。然后将原始输入连接起来,并作为单个源进行处理。请注意,mcDF中的JSON读取器接受源作为文件路径、原始字符串或类似文件的对象,以及这些源的可迭代对象。
>>> j1 = '{"id":0}\n{"id":1}\n'
>>> j2 = '{"id":2}\n{"id":3}\n'
>>> df = cudf.read_json([j1, j2], lines=True)
3.4.3. 展开列表和结构类型的数据
在将JSON数据读取到具有列表/结构列类型的mcDF数据帧中之后,许多工作流中的下一步会将数据提取或展平为简单类型。
对于结构列,一种解决方案是使用
struct.explode访问器提取数据,并将结果连接到父数据帧。以下示例演示如何从结构列中提取数据:>>> j = '\n'.join([ ... '{"x": "Tokyo", "y": {"country": "Japan", "iso2": "JP"}}', ... '{"x": "Jakarta", "y": {"country": "Indonesia", "iso2": "ID"}}', ... '{"x": "Shanghai", "y": {"country": "China", "iso2": "CN"}}' ... ]) >>> df = cudf.read_json(j, lines=True) >>> df = df.drop(columns='y').join(df['y'].struct.explode()) >>> df
x country iso2 0 Tokyo Japan JP 1 Jakarta Indonesia ID 2 Shanghai China CN
对于元素顺序有意义的列表列,
list.get访问器从特定位置提取元素。然后可以将生成的cudf.Series对象分配给数据帧中的一个新列。以下示例演示如何从列表列中提取第一个和第二个元素:>>> j = '\n'.join([ ... '{"name": "Peabody, MA", "coord": [42.53, -70.98]}', ... '{"name": "Northampton, MA", "coord": [42.32, -72.66]}', ... '{"name": "New Bedford, MA", "coord": [41.63, -70.93]}' ... ]) >>> df = cudf.read_json(j, lines=True) >>> df['latitude'] = df['coord'].list.get(0) >>> df['longitude'] = df['coord'].list.get(1) >>> df = df.drop(columns='coord') >>> df
name latitude longitude 0 Peabody, MA 42.53 -70.98 1 Northampton, MA 42.32 -72.66 2 New Bedford, MA 41.63 -70.93
对于长度可变的列表列,
explode方法将创建一个新的数据帧,每个元素都作为一行。将分解后的数据帧连接到父数据帧上会产生具有所有简单类型的输出。以下示例展平列表列,并将其连接到索引和父数据帧中的其他数据:>>> j = '\n'.join([ ... '{"product": "socks", "ratings": [2, 3, 4]}', ... '{"product": "shoes", "ratings": [5, 4, 5, 3]}', ... '{"product": "shirts", "ratings": [3, 4]}' ... ]) >>> df = cudf.read_json(j, lines=True) >>> df = df.drop(columns='ratings').join(df['ratings'].explode()) >>> df
product ratings 0 socks 2 0 socks 4 0 socks 3 1 shoes 5 1 shoes 5 1 shoes 4 1 shoes 3 2 shirts 3 2 shirts 4
3.4.4. 处理JSON数据
有时工作流需要使用对象根来处理JSON数据,mcDF提供了为此类数据构建解决方案的工具。如果需要使用对象根来处理JSON数据,建议将数据作为单个JSON行读取,然后拆包生成的数据帧。以下示例将JSON对象作为一行读取,然后将results字段提取到新的数据帧中:
>>> j = '''{
"metadata" : {"vehicle":"car"},
"results": [
{"id": 0, "distance": 1.2},
{"id": 1, "distance": 2.4},
{"id": 2, "distance": 1.7}
]
}'''
# first read the JSON object with line=True
>>> df = cudf.read_json(j, lines=True)
>>> df
metadata records
0 {'vehicle': 'car'} [{'id': 0, 'distance': 1.2}, {'id': 1, 'distan...
# then explode the 'records' column
>>> df = df['records'].explode().struct.explode()
>>> df
id distance
0 0 1.2
1 1 2.4
2 2 1.7
3.5. 缺失数据处理
在本节中,将讨论mcDF中缺失的(也称为NA)值。mcDF支持在所有dtype中都有缺失的值。这些缺失的值由<NA>表示,又称为“空值”。
3.5.1. 检测缺失数据
要检测缺失的值,可以使用 isna() 和 notna() 函数:
>>> import numpy as np
>>> import cudf
>>> df = cudf.DataFrame({"a": [1, 2, None, 4], "b": [0.1, None, 2.3, 17.17]})
>>> df
a b
0 1 0.1
1 2 <NA>
2 <NA> 2.3
3 4 17.17
>>> df.isna()
a b
0 False False
1 False True
2 True False
3 False False
>>> df["a"].notna()
0 True
1 True
2 False
3 True
Name: a, dtype: bool
必须注意的是,在Python(和NumPy)中,nan的比较不相等,但None的比较相等。请注意,mcDF/NumPy使用了np.nan != np.nan,并将None视为np.nan。
>>> None == None
True
>>> np.nan == np.nan
False
因此,与上面相比,标量与None/np.nan的比较并不能提供有用的信息。
>>> df["b"] == np.nan
0 False
1 <NA>
2 False
3 False
Name: b, dtype: bool
>>> s = cudf.Series([None, 1, 2])
s
0 <NA>
1 1
2 2
dtype: int64
>>> s == None
0 <NA>
1 <NA>
2 <NA>
dtype: bool
>>> s = cudf.Series([1, 2, np.nan], nan_as_null=False)
s
0 1.0
1 2.0
2 NaN
dtype: float64
>>> s == np.nan
0 False
1 False
2 False
dtype: bool
3.5.2. 浮点数据类型和缺失数据
因为NaN是一个浮点值,所以即使是一个整数数据类型的列,如果该列中有一个NaN,该列可能被强制转换为浮点数据类型。然而,默认不会出现这种情况。
默认情况下,如果将NaN值传递给Series构造函数,则将其视为<NA>值。
>>> cudf.Series([1, 2, np.nan])
0 1
1 2
2 <NA>
dtype: int64
因此,要将NaN视为NaN,必须将NaN_as_null=False参数传递到Series构造函数中。
>>> cudf.Series([1, 2, np.nan], nan_as_null=False)
0 1.0
1 2.0
2 NaN
dtype: float64
3.5.3. 日期数据类型
对于datetime64类型,mcDF不支持NaT值。相反,这些特定于NumPy和panda的值在mcDF中被视为null值(<NA>)。NaT的实际基础值是min(int64),在将mcDF对象转换为pandas对象时,mcDF保留基础值。
>>> import pandas as pd
>>> datetime_series = cudf.Series(
[pd.Timestamp("20120101"), pd.NaT, pd.Timestamp("20120101")]
)
>>> datetime_series
0 2012-01-01 00:00:00.000000
1 <NA>
2 2012-01-01 00:00:00.000000
dtype: datetime64[us]
>>> datetime_series.to_pandas()
0 2012-01-01
1 NaT
2 2012-01-01
dtype: datetime64[ns]
对datetime列中具有<NA>值的行执行的任何操作都将在结果列中的同一位置产生<NA>的值:
>>> datetime_series - datetime_series
0 0 days 00:00:00
1 <NA>
2 0 days 00:00:00
dtype: timedelta64[us]
3.5.4. 计算缺失数据
Null值通过pandas对象之间的算术运算自然传播:
>>> df1 = cudf.DataFrame(
{
"a": [1, None, 2, 3, None],
"b": cudf.Series([np.nan, 2, 3.2, 0.1, 1], nan_as_null=False),
}
)
>>> df2 = cudf.DataFrame(
{"a": [1, 11, 2, 34, 10], "b": cudf.Series([0.23, 22, 3.2, None, 1])}
)
>>> df1
a b
0 1 NaN
1 <NA> 2.0
2 2 3.2
3 3 0.1
4 <NA> 1.0
>>> df2
a b
0 1 0.23
1 11 22.0
2 2 3.2
3 34 <NA>
4 10 1.0
>>> df1 + df2
a b
0 2 NaN
1 <NA> 24.0
2 4 6.4
3 37 <NA>
4 <NA> 2.0
在对一系列数据求和时,NA值将被视为0:
>>> df1["a"]
0 1
1 <NA>
2 2
3 3
4 <NA>
Name: a, dtype: int64
>>> df1["a"].sum()
6
由于NA值被视为0,因此在这种情况下,平均值将为2(1+0+2+3+0)/5=2:
>>> df1["a"].mean()
2.0
为了在上述计算中保留NA值, sum 和 mean 支持 skipna 参数。默认情况下,它的值设置为True,可以将其更改为False以保留NA值:
>>> df1["a"].sum(skipna=False)
nan
>>> df1["a"].mean(skipna=False)
nan
累积方法(如 cumsum 和 cumprod )默认忽略NA值:
>>> df1["a"].cumsum()
0 1
1 <NA>
2 3
3 6
4 <NA>
Name: a, dtype: int64
要在累积方法中保留NA值,请提供 skipna=False:
>>> df1["a"].cumsum(skipna=False)
0 1
1 <NA>
2 <NA>
3 <NA>
4 <NA>
Name: a, dtype: int64
3.5.5. 对Null/NaNs进行点积和求和运算
数据帧的一个空系列或所有NA系列的总和为0:
>>> cudf.Series([np.nan], nan_as_null=False).sum()
0.0
>>> cudf.Series([np.nan], nan_as_null=False).sum(skipna=False)
nan
>>> cudf.Series([], dtype="float64").sum()
0.0
数据帧的空系列或所有NA系列的乘积为1:
>>> cudf.Series([np.nan], nan_as_null=False).prod()
1.0
>>> cudf.Series([np.nan], nan_as_null=False).prod(skipna=False)
nan
>>> cudf.Series([], dtype="float64").prod()
1.0
3.5.6. GroupBy操作中的NA数据
GroupBy中的NA组将自动排除在外。例如:
>>> df1
a b
0 1 NaN
1 <NA> 2.0
2 2 3.2
3 3 0.1
4 <NA> 1.0
>>> df1.groupby("a").mean()
b
a
2 3.2
1 NaN
3 0.1
也可以通过传递 dropna=False 将NA包括在组中:
>>> df1.groupby("a", dropna=False).mean()
b
a
2 3.2
1 NaN
3 0.1
<NA> 1.5
3.5.7. 插入缺失数据
所有数据类型都支持通过赋值插入缺少的值。通过将系列中的任何特定位置赋值为None,可以将其设为null:
>>> series = cudf.Series([1, 2, 3, 4])
>>> series
0 1
1 2
2 3
3 4
dtype: int64
>>> series[2] = None
>>> series
0 1
1 2
2 <NA>
3 4
dtype: int64
3.5.8. 使用fillna填充缺失数据
可以用非NA数据填充NA和NaN值:
>>> df1
a b
0 1 NaN
1 <NA> 2.0
2 2 3.2
3 3 0.1
4 <NA> 1.0
>>> df1["b"].fillna(10)
0 10.0
1 2.0
2 3.2
3 0.1
4 1.0
Name: b, dtype: float64
3.5.9. 使用mcDF对象填充缺失数据
也可以使用可对齐的dict或Series进行填充。dict的标签或Series的索引必须与要填充的帧的列相匹配。下列示例是用该列的平均值填充DataFrame:
>>> import cupy as cp
>>> dff = cudf.DataFrame(cp.random.randn(10, 3), columns=list("ABC"))
>>> dff.iloc[3:5, 0] = np.nan
>>> dff.iloc[4:6, 1] = np.nan
>>> dff.iloc[5:8, 2] = np.nan
>>> dff
A B C
0 0.819493 -0.944710 -0.937570
1 -1.150507 -0.051519 -1.250062
2 0.607780 -0.094671 -1.700941
3 NaN -0.025800 1.021424
4 NaN NaN -1.181323
5 -1.487396 NaN NaN
6 1.139386 0.991875 NaN
7 0.724027 -0.868154 NaN
8 0.803895 0.397944 -1.055434
9 -0.509395 0.647727 -1.731969
>>> dff.fillna(dff.mean())
A B C
0 0.819493 -0.944710 -0.937570
1 -1.150507 -0.051519 -1.250062
2 0.607780 -0.094671 -1.700941
3 0.118410 -0.025800 1.021424
4 0.118410 0.006587 -1.181323
5 -1.487396 0.006587 -0.976554
6 1.139386 0.991875 -0.976554
7 0.724027 -0.868154 -0.976554
8 0.803895 0.397944 -1.055434
9 -0.509395 0.647727 -1.731969
>>> dff.fillna(dff.mean()[1:3])
A B C
0 0.819493 -0.944710 -0.937570
1 -1.150507 -0.051519 -1.250062
2 0.607780 -0.094671 -1.700941
3 NaN -0.025800 1.021424
4 NaN 0.006587 -1.181323
5 -1.487396 0.006587 -0.976554
6 1.139386 0.991875 -0.976554
7 0.724027 -0.868154 -0.976554
8 0.803895 0.397944 -1.055434
9 -0.509395 0.647727 -1.731969
3.5.10. 丢弃缺失数据
可以使用 dropna() 排除丢失的数据:
>>> df1
a b
0 1 NaN
1 <NA> 2.0
2 2 3.2
3 3 0.1
4 <NA> 1.0
>>> df1.dropna(axis=0)
a b
2 2 3.2
3 3 0.1
>>> df1.dropna(axis=1)
0
1
2
3
4
Series有一个等效的 dropna():
>>> df1["a"].dropna()
0 1
2 2
3 3
Name: a, dtype: int64
3.5.11. 替换任意值
用其他值替换任意值。
Series中的 replace() 和DataFrame中的 replace() 提供了一种高效而灵活的方式来执行此类替换:
>>> series = cudf.Series([0.0, 1.0, 2.0, 3.0, 4.0])
>>> series
0 0.0
1 1.0
2 2.0
3 3.0
4 4.0
dtype: float64
>>> series.replace(0, 5)
0 5.0
1 1.0
2 2.0
3 3.0
4 4.0
dtype: float64
还可以将任何值替换为<NA>值:
>>> series.replace(0, None)
0 <NA>
1 1.0
2 2.0
3 3.0
4 4.0
dtype: float64
可以将值列表替换为其他值列表:
>>> series.replace([0, 1, 2, 3, 4], [4, 3, 2, 1, 0])
0 4.0
1 3.0
2 2.0
3 1.0
4 0.0
dtype: float64
还可以指定映射dict:
>>> series.replace({0: 10, 1: 100})
0 10.0
1 100.0
2 2.0
3 3.0
4 4.0
dtype: float64
对于DataFrame,可以按列指定各个值:
>>> df = cudf.DataFrame({"a": [0, 1, 2, 3, 4], "b": [5, 6, 7, 8, 9]})
>>> df
a b
0 0 5
1 1 6
2 2 7
3 3 8
4 4 9
>>> df.replace({"a": 0, "b": 5}, 100)
a b
0 100 100
1 1 6
2 2 7
3 3 8
4 4 9
3.5.12. 字符串/正则表达式替换
mcDF支持使用 replace API替换字符串值:
>>> d = {"a": list(range(4)), "b": list("ab.."), "c": ["a", "b", None, "d"]}
>>> df = cudf.DataFrame(d)
>>> df
a b c
0 0 a a
1 1 b b
2 2 . <NA>
3 3 . d
>>> df.replace(".", "A Dot")
a b c
0 0 a a
1 1 b b
2 2 A Dot <NA>
3 3 A Dot d
>>> df.replace([".", "b"], ["A Dot", None])
a b c
0 0 a a
1 1 <NA> <NA>
2 2 A Dot <NA>
3 3 A Dot d
替换几个不同的值(列表->列表):
>>> df.replace(["a", "."], ["b", "--"])
a b c
0 0 b b
1 1 b b
2 2 -- <NA>
3 3 -- d
仅在b列中搜索(dict -> dict):
>>> df.replace({"b": "."}, {"b": "replacement value"})
a b c
0 0 a a
1 1 b b
2 2 replacement value <NA>
3 3 replacement value d
3.5.13. 数值替换
可以参考 fillna() 的用法使用 replace() :
>>> df = cudf.DataFrame(cp.random.randn(10, 2))
>>> df[np.random.rand(df.shape[0]) > 0.5] = 1.5
>>> df.replace(1.5, None)
0 1
0 <NA> <NA>
1 <NA> <NA>
2 <NA> <NA>
3 -0.553375984 -1.008028523
4 <NA> <NA>
5 -1.65642176 -0.380188625
6 <NA> <NA>
7 1.661726764 -1.442556754
8 <NA> <NA>
9 <NA> <NA>
通过传递列表可以替换多个值:
>>> df00 = df.iloc[0, 0]
>>> df.replace([1.5, df00], [5, 10])
0 1
0 10.000000 10.000000
1 10.000000 10.000000
2 10.000000 10.000000
3 -0.553376 -1.008029
4 10.000000 10.000000
5 -1.656422 -0.380189
6 10.000000 10.000000
7 1.661727 -1.442557
8 10.000000 10.000000
9 10.000000 10.000000
也可以在DataFrame上就地操作:
>>> df.replace(1.5, None, inplace=True)
>>> df
0 1
0 <NA> <NA>
1 <NA> <NA>
2 <NA> <NA>
3 -0.553375984 -1.008028523
4 <NA> <NA>
5 -1.65642176 -0.380188625
6 <NA> <NA>
7 1.661726764 -1.442556754
8 <NA> <NA>
9 <NA> <NA>
3.6. GroupBy操作
mcDF支持pandas groupby API中的一小部分比较重要的API。
3.6.1. 支持的GroupBy操作列表
3.6.2. 分组
GroupBy对象是通过将Series或DataFrame的值按一列或多列分组来创建的:
>>> import cudf
>>> df = cudf.DataFrame({'a': [1, 1, 1, 2, 2], 'b': [1, 1, 2, 2, 3], 'c': [1, 2, 3, 4, 5]})
>>> df
a b c
0 1 1 1
1 1 1 2
2 1 2 3
3 2 2 4
4 2 3 5
>>> gb1 = df.groupby('a') # grouping by a single column
>>> gb2 = df.groupby(['a', 'b']) # grouping by multiple columns
>>> gb3 = df.groupby(cudf.Series(['a', 'a', 'b', 'b', 'b'])) # grouping by an external column
与pandas不同,mcDF默认情况下使用 sort=False 来获得更好的性能,这并不保证结果中有任何特定的组顺序。
例如:
>>> df = cudf.DataFrame({'a' : [2, 2, 1], 'b' : [42, 21, 11]})
>>> df.groupby('a').sum()
b
a
2 63
1 11
>>> df.to_pandas().groupby('a').sum()
b
a
1 11
2 63
设置 sort=True 将产生类似pandas的输出,但会带来一些性能损失:
>>> df.groupby('a', sort=True).sum()
b
a
1 11
2 63
3.6.2.1. 基于索引层分组
也可以按多索引的一个或多个级别进行分组:
>>> df = cudf.DataFrame(
... {'a': [1, 1, 1, 2, 2], 'b': [1, 1, 2, 2, 3], 'c': [1, 2, 3, 4, 5]}
... ).set_index(['a', 'b'])
...
>>> df.groupby(level='a')
3.6.2.2. Grouper对象
当列和级别具有相同名称时,Grouper可用于消除它们之间的歧义:
>>> df
b c
b
1 1 1
1 1 2
1 2 3
2 2 4
2 3 5
>>> df.groupby('b', level='b') # ValueError: Cannot specify both by and level
>>> df.groupby([cudf.Grouper(key='b'), cudf.Grouper(level='b')]) # OK
3.6.3. 聚合
通过 agg 方法支持对组进行聚合:
>>> df
a b c
0 1 1 1
1 1 1 2
2 1 2 3
3 2 2 4
4 2 3 5
>>> df.groupby('a').agg('sum')
b c
a
1 4 6
2 5 9
>>> df.groupby('a').agg({'b': ['sum', 'min'], 'c': 'mean'})
b c
sum min mean
a
1 4 1 2.0
2 5 2 4.5
>>> df.groupby("a").corr(method="pearson")
b c
a
1 b 1.000000 0.866025
c 0.866025 1.000000
2 b 1.000000 1.000000
c 1.000000 1.000000
表 3.2 总结了可用的聚合以及支持这些聚合的类型:
Agg/dtypes |
Numeric |
Datatime |
string |
Categorical |
List |
struct |
Interval |
Deceimal |
|---|---|---|---|---|---|---|---|---|
count |
Y |
Y |
Y |
Y |
Y |
|||
Size |
Y |
Y |
Y |
Y |
Y |
|||
Sum |
Y |
Y |
Y |
|||||
Idxmin |
Y |
Y |
Y |
|||||
Idxmax |
Y |
Y |
Y |
|||||
Min |
Y |
Y |
Y |
Y |
||||
max |
Y |
Y |
Y |
Y |
||||
mean |
Y |
Y |
||||||
var |
Y |
Y |
||||||
std |
Y |
Y |
||||||
quantile |
Y |
Y |
||||||
median |
Y |
Y |
||||||
nunique |
Y |
Y |
Y |
|||||
nth |
Y |
Y |
Y |
Y |
Y |
|||
collect |
Y |
Y |
Y |
Y |
||||
Unique |
Y |
Y |
Y |
Y |
||||
corr |
Y |
Y |
Y |
Y |
Y |
|||
cov |
Y |
Y |
3.6.4. 应用函数
要在每个组上应用函数,请使用 GroupBy.apply() 方法:
>>> df
a b c
0 1 1 1
1 1 1 2
2 1 2 3
3 2 2 4
4 2 3 5
>>> df.groupby('a').apply(lambda x: x.max() - x.min())
a b c
a
0 0 1 2
1 0 1 1
限制说明
apply实现方式是通过将提供的函数依次应用于每组,并将结果连接在一起。这可能非常缓慢,尤其是对于大量的小分组来说。对于少数大型组,它可以提供可接受的性能。
结果可能并不总是与pandas完全匹配。例如,mcDF可能返回一个包含单个column的DataFrame,而pandas返回Series。可能需要进行一些后处理以匹配pandas的行为。
mcDF不支持pandas apply支持的一些特殊情况,例如调用describe。
3.6.5. 转换
transform() 方法聚合每个组,并将结果广播到组大小,从而生成与输入Series/DataFrame大小相同的Series/Data Frame:
>>> import cudf
>>> df = cudf.DataFrame({'a': [2, 1, 1, 2, 2], 'b': [1, 2, 3, 4, 5]})
>>> df.groupby('a').transform('max')
b
0 5
1 3
2 3
3 5
4 5
3.6.6. 滑窗计算
使用 GroupBy.rolling() 方法对每个组执行滑窗计算:
>>> df
a b c
0 1 1 1
1 1 1 2
2 1 2 3
3 2 2 4
4 2 3 5
在每个分组上使用size为2的窗口进行求和:
>>> df.groupby('a').rolling(2).sum()
a b c
a
1 0 <NA> <NA> <NA>
1 2 2 3
2 2 3 5
2 3 <NA> <NA> <NA>
4 4 5 9
3.7. mcDF和mcPy互操作
本章节提供一些示例,介绍如何基于mcPy array功能(如高级线性代数运算)将mcDF和mcPy联合使用。
import timeit
import cupy as cp
from packaging import version
import cudf
if version.parse(cp.__version__) >= version.parse("10.0.0"):
cupy_from_dlpack = cp.from_dlpack
else:
cupy_from_dlpack = cp.fromDlpack
3.7.1. 将mcDF数据帧转换为mcPy数组
如果需要将mcDF DataFrame转换为mcPy ndarray,有多种方法可以实现:
使用
dlpack接口使用
DataFrame.values使用mcDF的
to_cupy功能进行转换
nelem = 10000
df = cudf.DataFrame(
{
"a": range(nelem),
"b": range(500, nelem + 500),
"c": range(1000, nelem + 1000),
}
)
%timeit arr_cupy = cupy_from_dlpack(df.to_dlpack())
%timeit arr_cupy = df.values
%timeit arr_cupy = df.to_cupy()
218 µs ± 5.62 µs per loop (mean ± std. dev. of 7 runs, 1,000 loops each)
687 µs ± 963 ns per loop (mean ± std. dev. of 7 runs, 1,000 loops each)
690 µs ± 2.22 µs per loop (mean ± std. dev. of 7 runs, 1,000 loops each)
arr_cupy = cupy_from_dlpack(df.to_dlpack())
arr_cupy
array([[ 0, 500, 1000],
[ 1, 501, 1001],
[ 2, 502, 1002],
...,
[ 9997, 10497, 10997],
[ 9998, 10498, 10998],
[ 9999, 10499, 10999]])
3.7.2. 将mcDF序列转换为mcPy数组
有多种方法可以将mcDF series转换为mcPy array:
将Series传递给
cupy.asarray使用
to_dlpack()使用
Series.values
col = "a"
%timeit cola_cupy = cp.asarray(df[col])
%timeit cola_cupy = cupy_from_dlpack(df[col].to_dlpack())
%timeit cola_cupy = df[col].values
103 µs ± 5.11 µs per loop (mean ± std. dev. of 7 runs, 10,000 loops each)
228 µs ± 2.2 µs per loop (mean ± std. dev. of 7 runs, 1,000 loops each)
210 µs ± 893 ns per loop (mean ± std. dev. of 7 runs, 1,000 loops each)
cola_cupy = cp.asarray(df[col])
cola_cupy
array([ 0, 1, 2, ..., 9997, 9998, 9999])
从这里开始,可以继续进行正常的mcPy工作流,例如reshaping数组、获取对角线或计算norm值:
reshaped_arr = cola_cupy.reshape(50, 200)
reshaped_arr
array([[ 0, 1, 2, ..., 197, 198, 199],
[ 200, 201, 202, ..., 397, 398, 399],
[ 400, 401, 402, ..., 597, 598, 599],
...,
[9400, 9401, 9402, ..., 9597, 9598, 9599],
[9600, 9601, 9602, ..., 9797, 9798, 9799],
[9800, 9801, 9802, ..., 9997, 9998, 9999]])
reshaped_arr.diagonal()
array([ 0, 201, 402, 603, 804, 1005, 1206, 1407, 1608, 1809, 2010,
2211, 2412, 2613, 2814, 3015, 3216, 3417, 3618, 3819, 4020, 4221,
4422, 4623, 4824, 5025, 5226, 5427, 5628, 5829, 6030, 6231, 6432,
6633, 6834, 7035, 7236, 7437, 7638, 7839, 8040, 8241, 8442, 8643,
8844, 9045, 9246, 9447, 9648, 9849])
cp.linalg.norm(reshaped_arr)
array(577306.967739)
3.7.3. 将mcPy数组转换为mcDF数据帧
可以将mcPy ndarray转换为mcDF DataFrame。有多种方法可以实现:
最简单的办法是直接使用DataFrame的构造函数
使用dlpack接口
对于后一种情况,需要确保mcPy数组在内存中是Fortran连续的。可以转置数组,也可以简单地预先强制它为Fortran连续数组。
%timeit reshaped_df = cudf.DataFrame(reshaped_arr)
34 ms ± 105 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
reshaped_df = cudf.DataFrame(reshaped_arr)
reshaped_df.head()
可以通过使用 cupy.isfortran 或查看array对象的 flags 属性来检查数组是否是Fortran连续的。
>>> cp.isfortran(reshaped_arr)
False
在这种情况下,需要在转到mcDF DataFrame之前对其进行转换。在接下来的示例中利用dlpack创建DataFrame。
%%timeit
fortran_arr = cp.asfortranarray(reshaped_arr)
reshaped_df = cudf.DataFrame(fortran_arr)
13.5 ms ± 119 µs per loop (mean ± std. dev. of 7 runs, 1 loop each)
%%timeit
fortran_arr = cp.asfortranarray(reshaped_arr)
reshaped_df = cudf.from_dlpack(fortran_arr.toDlpack())
9.49 ms ± 50.3 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
fortran_arr = cp.asfortranarray(reshaped_arr)
reshaped_df = cudf.DataFrame(fortran_arr)
reshaped_df.head()
3.7.4. 将mcPy数组转换为mcDF序列
要将数组转换为Series,可以直接将数组传递给Series构造函数:
>>> cudf.Series(reshaped_arr.diagonal()).head()
0 0
1 201
2 402
3 603
4 804
dtype: int64
3.7.5. 混合使用mcDF和mcPy构建平滑PyData工作流
mcDF正在快速发展,有时一个库可能不具备所需的功能。例如采用pandas DataFrame的逐行求和(或平均值)。mcDF对逐行操作的支持需要转换DataFrame或编写一个UDF,并显式计算每行的总和。根据数据的形状,转换可能会导致数十万列(mcDF不会很好地处理这些列),而编写UDF可能会耗费大量时间。
通过利用GPU PyData生态系统的互操作性,此操作变得非常简单。对之前reshaped的mcDF数据帧进行逐行求和。
reshaped_df.head()
可以将其转换为mcPy数组,并使用sum的axis参数:
>>> new_arr = cupy_from_dlpack(reshaped_df.to_dlpack())
>>> new_arr.sum(axis=1)
array([ 19900, 59900, 99900, 139900, 179900, 219900, 259900,
299900, 339900, 379900, 419900, 459900, 499900, 539900,
579900, 619900, 659900, 699900, 739900, 779900, 819900,
859900, 899900, 939900, 979900, 1019900, 1059900, 1099900,
1139900, 1179900, 1219900, 1259900, 1299900, 1339900, 1379900,
1419900, 1459900, 1499900, 1539900, 1579900, 1619900, 1659900,
1699900, 1739900, 1779900, 1819900, 1859900, 1899900, 1939900,
1979900])
只需这一行,就可以在这个生态系统中的数据结构之间无缝移动,在不牺牲速度的情况下提供巨大的灵活性。
3.7.6. 将mcDF数据帧转换为mcPy稀疏矩阵
可以将DataFrame或Series转换为mcPy稀疏矩阵。如果下游过程期望mcPy稀疏矩阵作为输入,可以这样做。
稀疏矩阵数据结构由三个密集array定义。以下将定义一个辅助函数用于精简代码:
def cudf_to_cupy_sparse_matrix(data, sparseformat="column"):
"""Converts an mcDF object to an mcPy Sparse Column matrix."""
if sparseformat not in (
"row",
"column",
):
raise ValueError("Let's focus on column and row formats for now.")
_sparse_constructor = cp.sparse.csc_matrix
if sparseformat == "row":
_sparse_constructor = cp.sparse.csr_matrix
return _sparse_constructor(cupy_from_dlpack(data.to_dlpack()))
可以定义一个稀疏填充的DataFrame来展示这种向稀疏矩阵格式的转换:
df = cudf.DataFrame()
nelem = 10000
nonzero = 1000
for i in range(20):
arr = cp.random.normal(5, 5, nelem)
arr[cp.random.choice(arr.shape[0], nelem - nonzero, replace=False)] = 0
df["a" + str(i)] = arr
df.head()
sparse_data = cudf_to_cupy_sparse_matrix(df)
print(sparse_data)
---------------------------------------------------------------------------
RuntimeError Traceback (most recent call last)
Cell In[21], line 2
1 sparse_data = cudf_to_cupy_sparse_matrix(df)
----> 2 print(sparse_data)
File /opt/conda/envs/docs/lib/python3.10/site-packages/cupyx/scipy/sparse/_base.py:61, in spmatrix.__str__(self)
58 def __str__(self):
59 # TODO(unno): Do not use get method which is only available when scipy
60 # is installed.
---> 61 return str(self.get())
File /opt/conda/envs/docs/lib/python3.10/site-packages/cupyx/scipy/sparse/_csc.py:64, in csc_matrix.get(self, stream)
50 """Returns a copy of the array on host memory.
51
52 .. warning::
(...)
61
62 """
63 if not _scipy_available:
---> 64 raise RuntimeError('scipy is not available')
65 data = self.data.get(stream)
66 indices = self.indices.get(stream)
RuntimeError: scipy is not available
从这里开始,可以使用mcPy稀疏矩阵继续工作流程。
3.8. pandas兼容性说明
DataFrame.quantile
与pandas的一个显著区别是,当DataFrame是非数字类型时,如果在pandas下计算结果是一个Series,那么mcDF将返回一个DataFrame,因为mcDF不支持混合数据类型的Series。
DataFrame.reindex, Series.reindex
与pandas的一个区别是,NA用于不匹配的行,而不是NaN。因为上述原因,如果reindex的结果列中具有不匹配的行,那么在mcDF中该列为整数数据类型,在pandas中它被强制转换为浮点。
DataFrame.truncate, Series.truncate
copy参数仅用于API兼容性,但不支持 copy=False。此方法总是生成一个副本。
Where
请注意,where将缺失的值视为false,与之对应的,pandas将其视为null数据:
>>> gsr = cudf.Series([1, 2, 3])
>>> gsr.where([True, False, cudf.NA])
0 1
1 <NA>
2 <NA>
dtype: int64
>>> gsr.where([True, False, False])
0 1
1 <NA>
2 <NA>
dtype: int64
MultiIndex.get_loc
此函数的返回类型可能与pandas提供的方法不同。如果索引既不是按字典排序的,也不是唯一的,则会尽最大努力将找到的索引强制转换为切片。例如:
>>> import pandas as pd
>>> import cudf
>>> x = pd.MultiIndex.from_tuples([
... (2, 1, 1), (1, 2, 3), (1, 2, 1),
... (1, 1, 1), (1, 1, 1), (2, 2, 1),
... ])
>>> x.get_loc(1)
array([False, True, True, True, True, False])
>>> cudf.from_pandas(x).get_loc(1)
slice(1, 5, 1)
groupby.apply
在某些情况下,pandas将分组键作为索引的一部分返回,而mcDF由于冗余而不返回。例如:
>>> import pandas as pd
>>> df = pd.DataFrame({
... 'a': [1, 1, 2, 2],
... 'b': [1, 2, 1, 2],
... 'c': [1, 2, 3, 4],
... })
>>> gdf = cudf.from_pandas(df)
>>> df.groupby('a').apply(lambda x: x.iloc[[0]])
a b c
a
1 0 1 1 1
2 2 2 1 3
>>> gdf.groupby('a').apply(lambda x: x.iloc[[0]])
a b c
0 1 1 1
2 2 1 3
3.9. 写时复制
写时复制(Copy-on-write)是一种内存管理策略,它允许包含相同数据的多个mcDF对象引用相同的内存地址,只要它们都不修改底层数据。 使用这种方法,任何生成对象未修改视图的操作(如副本、切片或DataFrame.head等方法)都会返回一个新对象,该对象指向与原始对象相同的内存。 但是,当修改现有对象或新对象时,会在修改之前复制数据,以确保更改不会在两个对象之间传播。
mcDF中的默认行为是禁用写时复制,因此要使用它,必须通过设置mcDF选项明确使能。 建议在脚本执行开始时设置写时复制,因为当在脚本执行过程中更改此设置时,将出现不可预期的行为,即启用写时复制时创建的对象仍将具有写时复制行为,而禁用写时复制时所创建的对象将具有不同的行为。
3.9.1. 启用写时复制
使用
cudf.set_option:>>> import cudf >>> cudf.set_option("copy_on_write", True)
在启动Python解释器之前,将环境变量
CUDF_COPY_ON_WRITE设置为1:export CUDF_COPY_ON_WRITE="1" python -c "import cudf"
3.9.2. 禁用写时复制
通过将 copy_on_write 选项设置为False,可以禁用写时复制:
>>> cudf.set_option("copy_on_write", False)
3.9.3. 执行复制
代码中不需要进行额外的更改即可使用写时复制:
>>> series = cudf.Series([1, 2, 3, 4])
执行浅层复制将创建一个指向相同底层设备内存的新Series对象:
>>> copied_series = series.copy(deep=False)
>>> series
0 1
1 2
2 3
3 4
dtype: int64
>>> copied_series
0 1
1 2
2 3
3 4
dtype: int64
当对系列或 copied_series 执行写入操作时,将创建数据的真实物理副本:
>>> series[0:2] = 10
>>> series
0 10
1 10
2 3
3 4
dtype: int64
>>> copied_series
0 1
1 2
2 3
3 4
dtype: int64
3.9.4. 说明
当启用写时复制时,在切片或索引时不再有视图的概念。修改mcDF创建的视图将始终触发复制,并且不会修改原始对象。
写时复制会产生更加一致的复制语义。由于每个对象都是原始对象的副本,用户不再需要考虑何时可能会意外地进行修改。 这将带来操作之间的一致性,并在为mcDF和pandas启用写时复制时使两者的行为保持一致。以下是一个pandas和mcDF在未启用写时复制时行为不一致的示例:
>>> import pandas as pd
>>> s = pd.Series([1, 2, 3, 4, 5])
>>> s1 = s[0:2]
>>> s1[0] = 10
>>> s1
0 10
1 2
dtype: int64
>>> s
0 10
1 2
2 3
3 4
4 5
dtype: int64
>>> import cudf
>>> s = cudf.Series([1, 2, 3, 4, 5])
>>> s1 = s[0:2]
>>> s1[0] = 10
>>> s1
0 10
1 2
>>> s
0 1
1 2
2 3
3 4
4 5
dtype: int64
当启用写时复制时,上述不一致性得到解决:
>>> import pandas as pd
>>> pd.set_option("mode.copy_on_write", True)
>>> s = pd.Series([1, 2, 3, 4, 5])
>>> s1 = s[0:2]
>>> s1[0] = 10
>>> s1
0 10
1 2
dtype: int64
>>> s
0 1
1 2
2 3
3 4
4 5
dtype: int64
>>> import cudf
>>> cudf.set_option("copy_on_write", True)
>>> s = cudf.Series([1, 2, 3, 4, 5])
>>> s1 = s[0:2]
>>> s1[0] = 10
>>> s1
0 10
1 2
dtype: int64
>>> s
0 1
1 2
2 3
3 4
4 5
dtype: int64
3.9.4.1. 深度复制和浅复制比较
Copy-on-Write enabled |
Copy-on-Write disabled (default) |
|
|---|---|---|
|
生成一个真正的副本,并且更改不会传播到原始对象 |
生成一个真正的副本,并且更改不会传播到原始对象 |
|
内存在两个对象之间共享,但对一个对象的任何写入操作都将在执行写入之前触发真正的物理复制。因此,更改不会传播到原始对象 |
内存在两个对象之间共享,对其中一个对象执行的更改将传播到另一个对象 |
3.10. Dask-mcDF扩展功能
Dask-mcDF在需要的情况下扩展了Dask,允许其DataFrame分区使用mcDF GPU DataFrame,而不是pandas DataFrame进行处理。 这使得在GPU上利用mcDF的高性能数据处理能力,加速大规模数据处理任务。特别是当数据集太大,无法容纳在单个GPU内存中时,Dask-mcDF允许在分布式GPU环境中进行高性能的数据处理。
3.10.1. 使用Dask
Dask是一个并行和分布式计算库,可扩展现有的Python和PyData生态系统。
Dask-mcDF安装后会将自己注册为Dask的DataFrame后端。这意味着,在许多情况下,使用mcDF支持的DataFrame只需要对现有工作流程做少量改动。 最小的改动就是在Dask的配置中选择mcDF作为DataFrame后端,在Python中可以这样实现:
import dask
dask.config.set({"dataframe.backend": "cudf"})
或者也可以在运行代码前在环境中设置 DASK_DATAFRAME__BACKEND=cudf。
3.10.2. 从磁盘格式创建DataFrame
如果工作流程从磁盘格式创建Dask DataFrame(例如使用dask.dataframe.read_parquet()),那么设置后端就足以迁移工作流程。
例如,考虑从parquet中读取DataFrame:
import dask.dataframe as dd
df = dd.read_parquet("data.parquet", ...)
要获得以mcDF为后端的DataFrame,必须设置 dataframe.backend 配置选项:
import dask
import dask.dataframe as dd
dask.config.set({"dataframe.backend": "cudf"})
df = dd.read_parquet("data.parquet", ...)
这段代码将使用mcDF的GPU加速的parquet reader来读取数据分区。
3.10.3. 从内存格式创建DataFrame
如果内存中已经有一个DataFrame,并希望将其转换为以mcDF为后端的DataFrame,则有两种选择,这取决于数据帧是否已经是Dask DataFrame。
如果是个Dask DataFrame,那么可以调用 dask.dataframe.to_backend() 将mcdf作为执行后端。
如果是个pandas DataFrame,那么可以调用 dask.dataframe.from_pandas() 然后再调用 to_backend(),或者先用 cudf.from_pandas() 转换DataFrame,然后用 dask_cudf.from_cudf() 并行处理。
3.10.4. 分布式扩展Dask-mcDF
在使用Dask时,大多数情况下都会使用分布式调度程序,它存在于Dask集群中。Dask集群的结构如下图所示:
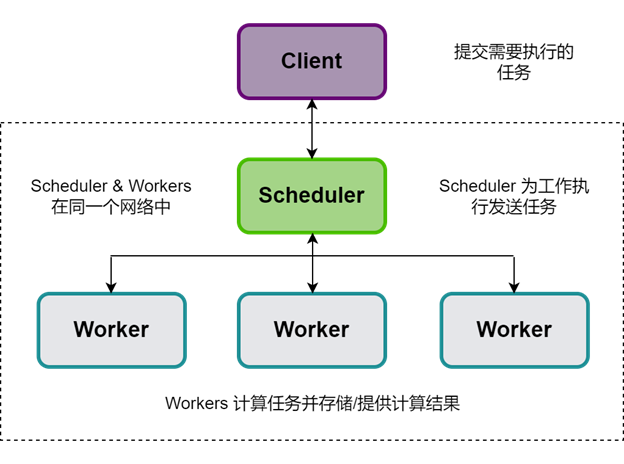
图 3.1 Dask集群结构
Dask-MACA是一个扩展了Dask.distributed的单机LocalCluster的库,可用于单机上分布式GPU工作负载, n_workers 参数适合接收单机上GPU数目的数值。
一旦创建好Dask-MACA实现的本地多GPU集群的实例cluster,就可以将Dask客户端附加到该集群上,并开始提交任务调度给多个GPU资源进行并行处理:
client = dask.distributed.Client(cluster)
以Kubernetes集群为例,GPU集群环境的部署方案,操作步骤如下:
制作Dask-mcDF基础容器镜像。
Dask-mcDF的基础容器镜像需要能运行mcDF和Dask-mcDF的完整功能,Dask集群中的scheduler和workers角色都将基于此基础镜像的Kubernetes资源中执行任务。
备注
在Docker容器中使用加速卡,参见《曦云® 系列通用计算GPU用户指南》。
利用Kubernetes资源定义的Chart文件,可用于部署Dask集群。将这个Helm Chart安装到Kubernetes集群中可以自动化部署Dask集群到Kubernetes集群环境中。
备注
Helm Chart支持参数化配置,允许在部署时定制应用程序的行为。 这些参数可以在Chart的values.yaml文件中定义,并通过
helm install命令的--set标志或-f标志(指向包含覆盖值的YAML文件)进行覆盖。获取Dask scheduler的url可实例化Dask集群管理对象,通过连接客户端到Dask集群,开始提交任务到Kubernetes提供的集群环境,Python代码如下:
client = dask.distributed.Client("dask_scheduler_url")
4. mcDF编程接口
mcDF的API接口兼容cuDF接口,详细信息可参见 cuDF API参考文档。
Dask-mcDF的API接口兼容Dask-cuDF接口,详细信息可参见 Dask-cuDF API参考文档。
5. 附录
5.1. 术语/缩略语
术语/缩略语 |
全称 |
说明 |
|---|---|---|
pandas |
一个开源的第三方Python库 |
|
Split-Apply-Combine |
拆分-应用-合并 |
|
I/O |
Input/Output |
输入/输出 |
MXMACA |
MetaX Advanced Compute Architecture |
沐曦推出的GPU软件栈,包含了沐曦GPU的底层驱动、编译器、数学库及整套软件工具套件 |
UDF |
User Defined Function |
用户定义函数 |